分子杂交
| A+医学百科 >> 分子杂交 |
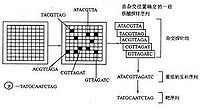
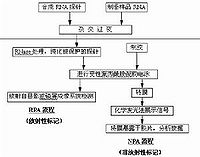
分子杂交(molecularhybridization)确定单链核酸碱基序列的技术。其基本原理是待测单链核酸与已知序列的单链核酸(叫做探针)间通过碱基配对形成可检出的双螺旋片段。这种技术可在DNA与DNA,RNA与RNA,或DNA与RNA之间进行,形成DNA-DNA,RNA-RNA或RNA-DNA等不同类型的杂交分子。作为探针的已知DNA或RNA片段一般为30~50核苷酸长,可用化学方法合成或者直接利用从特定细胞中提取的mRNA。探针必须预先标记以便检出杂交分子。标记方法有多种,常用的为同位素标记法和生物素标记法。杂交方法又可分为液相杂交和固相杂交。
使单链DNA或 RNA分子与具有互补碱基的另一DNA或RNA 片断结合成双链的技术。即核酸分子间的杂交。相互杂交的两种核酸分子间有时并非完全一致,但必有一定的相关性。早在1961年有人创建了DNA与RNA间的分子杂交,但至70年代才发展成基因分析中一种重要的分析技术,可鉴定基因的特异性。例如可将具有一定已知顺序的某基因的 DNA片段,标上放射性核素,构成核酸探针,将它通过分子杂交与缺陷的基因结合,产生杂交信号,从而把缺陷的基因显示出来,借此可对许多遗传性疾病进行产前诊断。现又用核酸分子杂交技术诊断乙型肝炎,以及研究其他病毒性疾病和癌瘤的基因结构(癌基因)等。
核酸分子杂交的方法并不复杂。在杂交前先把待分析的DNA加热或加碱使之变性,分开成单链;然后加入放射性核素标记的核酸探针,降温退火,即获带有放射性核素标记的双链杂交分子。1979年又发展成转膜杂交,即将电泳分开的核酸片段,经过转移电泳转移到硝酸纤维素膜上,进行固相杂交反应,用放射自显影检出之。此即著名的萨瑟恩氏印迹法。
无庸置疑,分子杂交的基础是两个核酸分子间的完全一致,或有一定的相似性或相关性。若所用的核酸探针的片段较长(几百个核苷酸以上),可以得到很准确的鉴定结果。但若人工合成的寡核苷酸探针片段较短(如20~30个核苷酸),则难免会出现准确性差的假阳性现象。现又有非放射性核素标记核酸探针,如以碱性磷酸酶标记的酶标核酸探针,以及以生物素标记的核酸探针等,这必将有助于推动分子杂交在临床医学中的广泛应用。
目录 |
杂交技术
目前使用较多的是固相杂交法。此法是先将待测单链核酸样品(如为双链,则须先变性成为单链)结合到硝酸纤维素膜上,然后与溶液中的标记探针进行杂交。通过与电泳法和放射自显影法结合,获得杂交图谱,再进行定性或定量分析。分子杂交方法广泛用于生物化学、分子生物学中作为核酸片段碱基序列的检测与鉴定手段。在医学领域中已用于某些病毒或细菌引起的感染性疾病的诊断。它也可用于基因工程。不同来源蛋白质的亚基结合过程也可称为杂交。
分子杂交(molecularhybridization)不同来源的核酸单链之间或蛋白质亚基之间由于结构互补而发生的非共价键的结合。根据这一原理发展起来的各种技术统称为分子杂交技术,核酸分子杂交技术是分子遗传学中的重要研究方法。
核酸分子杂交具有互补的碱基顺序的单链核酸分子,可以通过碱基对之间非共价键(主要是氢键)的形成即能出现稳定的双链区,这是核酸分子杂交的基础。杂种分子的形成并不要求两条单链的碱基顺序完全互补,所以不同来源的核酸单链只要彼此之间有一定程度的互补顺序(即某种程度的同源性)就可以形成杂种双链。分子杂交可在DNA与DNA、RNA与RNA或RNA与DNA的二条单链之间进行。由于DNA一般都以双链形式存在,因此在进行分子杂交时,应先将双链DNA分子解聚成为单链,这一过程称为变性,一般通过加热或提高pH值来实现。使单链聚合为双链的过程称为退火或复性。用分子杂交进行定性或定量分析的最有效方法是将一种核酸单链用同位素标记成为探针,再与另一种核酸单链进行分子杂交。由于同位素被检出的灵敏度高,所以在两种核酸分子之间即使只有百万分的同源顺序也可以彼检出。
核酸分子杂交按杂交中单链核酸所处的状态可分为液相、固相和原位3类。前二类方法都要求预先从细胞中分离并纯化杂交用的核酸。在液相分子杂交中,两种来源的核酸分子都处于溶液中,可以自由运动,其中有一种常是用同位素标记的。从复性动力学数据的分析可探知真核生物基因组结构的大致情况,如各类重复顺序的含量及分布情况等。在固相分子杂交中,一种核酸分子被固定在不溶性的介质上,另一种核酸分子则处在溶液中,两种介质中的核酸分子可以自由接触。常用的介质有硝酸纤维素滤膜、羟基磷灰石柱、琼脂和聚丙烯酰胺凝胶等。早期用琼脂作为固定介质的分子杂交方法曾被用来测定从细菌到人多种生物的DNA的同源程度。
1975年由E.萨瑟恩首创的萨瑟恩吸印法是一种方便易行的固相杂交方法。先将待测的DNA用限制性内切酶切成片段,然后通过凝胶电泳把大小不同的片段分开,再把这些DNA片段吸印到硝酸纤维膜上,并使吸附在滤膜上的DNA分子变性,然后和预先制备的DNA或RNA探针进行分子杂交,最后通过放射自显影就可以鉴别待测的哪个DNA片段和探针具有同源顺序。精确控制杂交及洗涤条件(如温度及盐浓度),在同源顺序中即使有一对碱基错配也可检出。这技术已应用于遗传病(基因病)的临床诊断。另一种相似的方法是将待测的RNA片段吸印在重氮苄氧甲基(DBM)纤维素膜或滤纸上,然后和预先制备的DNA探针进行分子杂交,这种方法称为诺森吸印法。这些技术大大地推进了分子遗传学研究和重组DNA技术的应用。
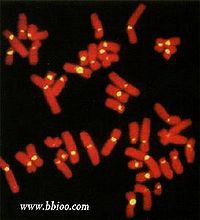
原位(分子)杂交实际上是固相杂交的另一种形式,杂交中的一种DNA处在未经抽提的染色体上,并在原来位置上被变性成单链,再和探针进行分子杂交。在原位杂交中所使用的探针必须用比活性高的同位素标记。杂交的结果可用放射自显影来显示,出现银粒的地方就是与探针互补的顺序所在的位置。
基因定位
在基因定位的工作中,对固定在细胞学制片上的染色体DNA进行原位杂交,可以将与探针互补的顺序精确地定位到染色体的一定位置上。
在基因定位工作中,还可以应用一种在电镜下直接观察杂种分子的原位杂交方法,即所谓的R-环法。R-环是在双链DNA分子部分变性的情况下,单链的RNA分子和相应的DNA序列中的一条互补的单链形成RNA-DNA的杂合双链,同时DNA的另一条单链向外突出而形成的环状图象。由于每种mRNA由特定的基因所转录,和这一基因的一个DNA单链具有互补结构,因此通过观察标记mRNA参与构成的R环的位置就可以对产生这种mRNA的基因进行定位,这一方法还可以用来研究断裂基因的结构。
在重组DNA技术中,有些专门的原位杂交方法可以用来筛选有探针顺序的重组质粒或噬菌体。此外,分子杂交方法还可以用来分离具有特定顺序的核酸分子。例如使探针与细胞的总DNA进行分子杂交,利用羟基磷灰石柱只吸附双链分子的特性,可以分离与探针互补的DNA片段或RNA分子。
蛋白质分子杂交来自不同的蛋白质的亚基间也可以形成非共价键的结合,这是蛋白质分子杂交的基础。蛋白质分子杂交技术可以用来研究蛋白质的结构和功能以及相似的蛋白质(如同工酶、血红蛋白等)的亲缘关系。例如通过人、狗、兔、驴、小鼠和蛙等动物的血红蛋白的蛋白质分子杂交测验,根据能否形成杂种分子和杂种分子的量,可以判断它们的蛋白质分子的相似程度。
实验原理
分子杂交是通过配对碱基对之间的非共价键(主要是氢键)结合,从而形成稳定的双链区。杂交分子的形成并不要求两条单链的碱基顺序完全互补,所以不同来源的核酸单链只要彼此之间有一定程度的互补顺序(即某种程度的同源性)就可以形成杂交双链。分子杂交可在DNA与DNA、RNA与RNA或RNA与DNA的二条单链之间进行。由于DNA一般都以双链形式存在,因此在进行分子杂交时,应先将双链DNA分子解聚成为单链,这一过程称为变性,一般通过加热或提高pH值来实现。用分子杂交进行定性或定量分析的最有效方法是将一种核酸单链用同位素或非同位素标记成为探针,再与另一种核酸单链进行分子杂交。
杂交类型
随着基因工程研究技术的迅猛发展,新的核酸分子杂交类型和方法在不断涌现和完善。核酸分子杂交可按作用环境大致分为固相杂交和液相杂交两种类型:
(1)固相杂交
将参加反应的一条核酸链先固定在固体支持物上,一条反应核酸游离在溶液中。固体支持物有硝酸纤维素滤膜、尼龙膜、乳胶颗粒、磁珠和微孔板等。由于固相杂交后,未杂交的游离片段可容易地漂洗除去,膜上留下的杂交物容易检测和能防止靶DNA自我复性等优点,所以最为常用。常用的固相杂交类型有:菌落原位杂交、斑点杂交、狭缝杂交、Southern印迹杂交、Northern印迹杂交、组织原位杂交和夹心杂交等。
(2)液相杂交
所参加反应的两条核酸链都游离在溶液中,一种研究最早且操作复合的杂交类型,在过去的30年里虽有时被应用,但总不如固相杂交那样普遍。主要缺点是杂交后过量的未杂交探针在溶液中除去较为困难和误差较高。近几年由于杂交检测技术的不断改进,商业性基因探针诊断盒的实际应用,推动了液相杂交技术的迅速发展。
固相膜核酸分子杂交
(1)菌落原位杂交(Colonyinsituhybridization)
将细菌从培养平板转移到硝酸纤维素滤膜上,然后将滤膜上的菌落裂菌以释出DNA,再烘干固定DNA于膜上,与32P标记的探针杂交,放射自显影检测菌落杂交信号,并与平板上的菌落对位。
(2)斑点杂交(Dotblotting)
该方法是将被检标本点到膜上,烘烤固定。这种方法耗时短,可做半定量分析,一张膜上可同时检测多个样品。为使点样准确方便,市售有多种多管吸印仪(manifolds),如MinifoldI和II、Bio-Dot(Bio-Rad)和Hybri-Dot,它们有许多孔,样品加到孔中,在负压下就会流到膜上呈斑点状或狭缝状,反复冲洗进样孔,取出膜烤干或紫外线照射以固定标本,这时的膜就可以进行杂交。
(3)Southern印迹杂交(Southernblotting)
是研究DNA图谱的基本技术,在遗传诊断DNA图谱分析及PCR产物分析等方面有重要价值。基本方法是将DNA标本用限制性内切酶消化后,经琼脂糖凝胶电泳分离各酶解片段,然后经碱变性,Tris缓冲液中和,高盐下通过毛吸作用将DNA从凝胶中转印至硝酸纤维素滤膜(尼龙膜也较长用)上,烘干固定后即可用于杂交。凝胶中DNA片段的相对位置转移到滤膜的过程中继续保持着。附着在滤膜上的DNA与32P标记的探针杂交,利用放射自显影术确定探针互补的每条DNA带的位置,从而可以确定在众多酶解产物中含某一特定序列的DNA片段的位置和大小。
(4)Northern印迹杂交(Northernblotting)
DNA印迹技术由Southern于1975年创建,称为Southern印迹技术。RNA印迹技术正好与DNA相对应,故被称为Northern印迹杂交,与此原理相似的蛋白质印迹技术则被称为westernblotting。Northern印迹杂交的RNA吸印与Southern印迹杂交的DNA吸印方法类似,只是在进样前用甲基氧化汞、乙二醛或甲醛使RNA变性,而不用NaOH,因为它会水解RNA的2’-羟基基团。RNA变性后有利于在转印过程中与硝酸纤维素膜结合,它同样可在高盐中进行转印,但在烘烤前与膜结合得并不牢固,所以在转印后不能用低盐缓冲液洗膜,否则RNA会被洗脱。在胶中不能加EB,因它为影响RNA与硝酸纤维素膜的结合,为测定片段大小,可在同一块胶上加标记物一同电泳,之后将标记物胶切下,上色、照像。样品胶则进行Northern转印,标记物胶上色的方法是在暗室中将其浸在含5μg/mlEB的0.1mol/L醋酸铵中10min,在水中就可脱色。在紫外光下用一次成像相机拍照时,上色的RNA胶要尽可能少接触紫外光,若接触太多或白炽灯下暴露过久,会使RNA信号降低。从琼脂糖凝胶中分离功能完整的mRNA时,甲基氢氧化汞是一种强力、可逆变性剂,但是有毒,因而许多人喜用甲醛作为变性剂。所有操作均应避免RNase的污染。
(5)组织原位杂交(Tissueinsituhybridization)
简称原位杂交,指组织或细胞的原位杂交,它与菌落的原位杂交不同,菌落原位杂交需裂解细菌释出DNA,然后进行杂交。而原位杂交是经适当处理后,使细胞通透性增加,让探针进入细胞内与DNA或RNA杂交。因此原位杂交可以确定探针的互补序列在胞内的空间位置,这一点具有重要的生物学和病理学意义。例如,对致密染色体DNA的原位杂交可用于显示特定的序列的位置;对分裂期间核DNA的杂交可研究特定序列在染色质内的功能排布;与细胞RNA的杂交可精确分析任何一种RNA在细胞中和组织中的分布。此外,原位杂交还是显示细胞亚群分布和动向及病原微生物存在方式和部位的一种重要技术。
用于原位杂交的探针可以是单链或双链DNA,也可以是RNA探针,探针的长度通常以100~400nt为宜,过长则杂交效率减低。最近研究结果表明,寡核苷酸探针(16~30nt)能自由出入细菌和组织细胞壁,杂交效率明显高于长探针。因此,寡核苷酸探针和不对称PCR标记的小DNA探针或体外转录标记的RNA探针是组织原位杂交的优选探针。
试验方法
杂交
通过碱基对之间非共价键的形成即出现稳定的双链区,这是核酸分子杂交的基础。杂交分子的形成并不要求两条单链的碱基顺序完全互补,所以不同来源的核酸单链只要彼此之间有一定程度的互补顺序(即某种程度的同源性)就可以形成杂交双链。分子杂交可在DNA与DNA、RNA与RNA或RNA与DNA的二条单链之间进行。由于DNA一般都以双链形式存在,因此在进行分子杂交时,应先将双链DNA分子解聚成为单链,这一过程称为变性,一般通过加热或提高pH值来实现。使单链聚合双链的过程称为退火或复性。用分子杂交进行定性或定量分析的最有效方法是将一种核酸单链用同位素或非同位素标记成为探针,再与另一种核酸单链进行分子杂交。核酸杂交技术基本上是Hall等1961年的工作开始的,探针与靶序列在溶液中杂交,通过平衡密度梯度离心分离杂交体。该法很慢、费力且不精确,但它开拓了核酸杂交技术的研究。Bolton等1962年设计了第一种简单的固相杂交方法,称为DNA-琼脂技术。变性DNA固定在琼脂中,DNA不能复性,但能与其它互补核酸序列杂交。典型的反应是用放射性标记的短DAN或RNA分子与胶中DNA杂交过夜,然后将胶置于柱中进行漂洗,去除游离探针,在高温、低盐条件下将结合的探针洗脱,洗脱液的放射性与结合的探针量呈正比。该法尤其适用于过量探针的饱和杂交实验。60年代末,Britten等设计了另一种分析细胞基因组的方法。该法是研究液相中DNA的复性以比较不同来源核酸的复杂度,典型的方法是:从不同生物体(细菌、酵母、鱼和哺乳动物等)内分离DNA,用水压器剪切成长约450核苷酸(nt)的片段。剪切的DNA液(含0.12mol/L磷酸盐缓冲液或0.18mol/LNa+),经煮沸使dsDNA热变性成ssDNA。然后冷至约60℃,在此温度孵育过程中,测定溶液一定时间内的UV260nm的吸光度(减色效应)来监测互补链的复性程度。通常该实验可比较不同来源生物DNA的复性速率,并可建立序列复杂度与动力学复杂度间的关系。
60年代中期Nygaard等的研究为应用标记DNA或RNA探针检测固定在硝酸纤维素(NC)膜上的DNA序列奠定了基础。如Brown等应用这一技术评估了爪蟾rRNA基因的拷贝数。RNA在代谢过程中被3H尿嘧啶标记,并在过量的情况下与膜上固定的基因组DNA杂交,继而用RNase处理,消化非特异性结合的RNA。漂洗后计数以测定杂交探针的量。通过计算与已知量DNA杂交的RNA量即可评估rRNA基因数。由于当时缺乏特异探针,这种方法不能用于研究其它特异基因的表达,这些早期过量探针膜杂交试验实际上是现代膜杂交实验的基础。进入70年代早期,许多重要的发展促进了核酸杂交技术的进展。例如,对特异基因转录产物的分析和对动力学杂交实验又有兴趣。固相化的PolyU–Sepharose和寡(dT)-纤维素使人们能从总RNA中分离PolyA+RNA。用mRNA的经纯化技术可从网织红细胞总RNA中制备α-和β-珠蛋白mRNA混合物。这些珠蛋白mRNA首次被用于合成特异的探针以分析珠蛋白基因的表达。由于制备cDNA探针很繁琐,所获得cDNA的长度和纯度也不稳定。所以寻求新的探针来源是使分子杂交技术进一步推广的基础。
70年代末期到80年代早期,分子生物学技术有了突破性进展,限制性内切酶的发展和应用使分子克隆成为可能。各种载体系统的诞生,尤其是质粒和噬菌体DAN载体的构建,使特异性DNA探针的来源变得十分丰富。人们可以从基因组DNA文库和cDNA文库中获得特定基因克隆,只需培养细菌,便可提取大量的探针DNA。迄今为止,已克隆和定性了许多特异DNA探针。
由于固相化学技术和核酸自动合成仪的诞生,现在可常规制备18~100个碱基的寡核苷酸探针。应用限制酶和Southern印迹技术,用数微克DNA就可分析特异基因。特异DNA或RNA序列的量和大小均可用Southern印迹和Northern印迹来测定,与以前的技术相比,大大提高了杂交水平和可信度。
尽管取得了上述重大进展,但分子杂交技术在临床实用中仍存在不少问题,必须提高检测单拷贝基因的敏感性,用非放射性物质代替放射性同位素标记探针以及简化实验操作和缩短杂交时间,这样,就需要在以下三方面着手研究:第一,完善非放射性标记探针;第二,靶序列和探针的扩增以及信号的放大;第三,发展简单的杂交方式,只有这样,才能使DNA探针实验做到简便、快速、低廉和安全。
探针-靶反应
从化学和生物学意义上理解,探针是一种分子,它带有供反应后检测的合适标记物,并仅与特异靶分子反应。抗原-抗体、外源凝集素-碳水化合物、亲和素-生物素、受体-配基(ligand)以及互补核酸间的杂交均属于探针-靶分子反应。蛋白质探针(如抗体)与特异靶分子是通过混合力(疏水、离子和氢键)的作用在少数特异位点上的结合,而核酸探针与互补链的反应则是根据杂交体的长短不同,通过氢键在几十、几百甚至上千个位点上的结合。因为有机溶液可降低杂交体的稳定性,所以,疏水反应对互补核酸链的结合也有一定的作用,但对其特异性影响甚微。
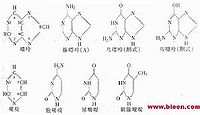
核苷酸经某一原子、功能基团或长侧链修饰后仍可能进行碱基配对,这取决于修饰的部位和修饰的性质。这一特性有助于理解非放射性核酸探针标记物的设计和125I与DNA探针的化学结合。能与核酸结合的单一原子有银、溴和碘等,这些元素可与嘧啶(胸腺嘧啶除外)环的C-5位或嘌呤环的C-8位反应而不影响氢键的形成。溴亦可与胸腺嘧啶的C-6位结合。而胞嘧啶的C-4和腺嘌呤的N-6就不能被修饰,否则会影响碱基配对,尽管C的N-4位和A的N-6位参与了氢键形成,但它们也是标记位点。这是因为标记的探针每1kb只掺入10~30个修饰碱基,即仅4%~12%的单个碱基被修饰的类似物取代了。尽管掺入位点处的碱基配对较弱或不存在,但对整个杂交分子的稳定性影响很小。防止氢键破坏的一种方法就是修饰探针,即探针克隆入M13载体中,只修饰载体区而不修饰插入片段。当用放射性同位素32P和35S标记核酸时,由于同位素是掺入核酸骨架的磷酸二脂键中,因此碱基未发生任何修饰。在5’端的磷酸基团上可进行化学修饰,这是标记寡核苷酸探针的有效方法。因为这种方法是在一个探针分子上标记一个检测的基团,所以,对长的克隆探针不适用。
此外,还可利用修饰的碱基来增加杂交的稳定性和特异性。2-氢基腺嘌呤可替代寡核苷酸探针中的腺嘌呤通过形成3个氢键以增加杂交体的稳定性。另外,在G-C丰富的RNA探针中,可用次黄嘌呤代替鸟嘌呤以获得特异的杂交。因为次黄嘌呤和鸟嘌呤间只形成2个氢键,有效地降低了杂交体的Tm值,这样,Tm值与杂交温度更接近,杂交的严格性就增加了,因此,也就增加了特异性。
很显然,结合位点的不同和可检测基团与检测系统的不同,可派生出很多核酸探针标记方法。这是由核酸的化学结构和性质所决定的。只有在对核酸分子的探针-靶反应的化学本质有了深入了解之后,才能更好地理解后面内容。
探针种类
分子杂交基因探针根据标记方法不同可粗分为放射性探针和非放射性探针两大类,根据探针的核酸性质不同又可分为DNA探针,RNA探针,cDNA探针,cRNA探针及寡核苷酸探针等几类,DNA探针还有单链和双链之分。下面分别介绍这几种探针。
(一)DNA探针
DNA探针是最常用的核酸探针,指长度在几百碱基对以上的双链DNA或单链DNA探针。现已获得DNA探针数量很多,有细菌、病毒、原虫、真菌、动物和人类细胞DNA探针。这类探针多为某一基因的全部或部分序列,或某一非编码序列。这些DNA片段须是特异的,如细菌的毒力因子基因探针和人类Alu探针。这些DNA探针的获得有赖于分子克隆技术的发展和应用。以细菌为例,目前分子杂交技术用于细菌的分类和菌种鉴定比之G+C百分比值要准确的多,是细菌分类学的一个发展方向。加之分子杂交技术的高敏感性,分子杂交在临床微生物诊断上具有广阔的前景。细菌的基因组大小约5×106bp,约含3000个基因。各种细菌之间绝大部分DNA是相同的,要获得某细菌特异的核酸探针,通常要采取建立细菌基因组DNA文库的办法,即将细菌DNA切成小片段后分别克隆得到包含基因组的全信息的克隆库。
然后用多种其它菌种的DNA作探针来筛选,产生杂交信号的克隆被剔除,最后剩下的不与任何其它细菌杂交的克隆则可能含有该细菌特异性DNA片段。将此重组质粒标记后作探针进一步鉴定,亦可经DNA序列分析鉴定其基因来源和功能。因此要得到一种特异性DNA探针,常常是比较繁琐的。探针DNA克隆的筛选也可采用血清学方法,所不同的是所建DNA文库为可表达性,克隆菌落或噬斑经裂解后释放出表达抗原,然后用来源细菌的多克隆抗血清筛选阳性克隆,所得到多个阳性克隆再经其它细菌的抗血清筛选,最后只与本细菌抗血清反应的表达克隆即含有此细菌的特异性基因片段,它所编码的蛋白是该菌种所特有的。用这种表达文库筛选得到的显然只是特定基因探针。
对于基因探针的克隆尚有更快捷的途径。这也是许多重要蛋白质的编码基因的克隆方法。该方法的第一步是分离纯化蛋白质,然后测定该蛋白的氨基或羟基末端的部分氨基酸序列,然后根据这一序列合成一套寡核苷酸探针。用此探针在DNA文库中筛选,阳性克隆即是目标蛋白的编码基因。值得一提的是真核细胞和原核细胞DNA组织有所不同。真核基因中含有非编码的内含子序列,而原核则没有。因此,真核基因组DNA探针用于检测基因表达时杂交效率要明显低于cDNA探针。DNA探针(包括cDNA探针)的主要优点有下面三点:①这类探针多克隆在质粒载体中,可以无限繁殖,取之不尽,制备方法简便。②DNA探针不易降解(相对RNA而言),一般能有效抑制DNA酶活性。③DNA探针的标记方法较成熟,有多种方法可供选择,如缺口平移,随机引物法,PCR标记法等,能用于同位素和非同位素标记。
(二)cDNA探针
cDNA(complementaryDNA)是指互补于mRNA的DNA分子。cDNA是由RNA经一种称为逆转录酶(reversetranscriptase)的DNA聚合酶催化产生的,这种逆录酶是Temin等在70年代初研究致癌RNA病毒时发现的。该酶以RNA为模板,根据碱基配对原则,按照RNA的核苷酸顺序合成DNA(其中U与A配对)。这一途径与一般遗传信息流的方向相反,故称反向转录或逆转录。携带逆转录酶的病毒侵入宿主细胞后,病毒RNA在逆转录酶的催化下转化成双链cDNA,并进而整合人宿主细胞染色体DNA分子,随宿主细胞DNA复制同时复制。这种整合的病毒基因组称为原病毒。在静止状态下,可被复制多代,但不被表达,故无毒性。一旦因某种因素刺激而被活化,则该病毒大量复制,如其带有癌基因,还可能诱发细胞癌变,后来发现逆转录酶不仅普遍存在于RNA病毒中,而且哺乳动物的胚胎细胞和正在分裂的淋巴细胞也含有逆转录酶。逆转录酶的作用是以dNTP为底物,RNA为模板,tRNA(主要是色氨酸tRNA)为引物,在Trna3’-OH末端上,5’-3’方向,合成与RNA互补的DNA单链,称为互补DNA(cDNA),单链cDNA与模板RNA形成RNA-DNA杂交体。随后在逆转录酶的RNaseH活性作用下,将RNA链水解成小片段。cDNA单链的3’末端回折形成一个小引物末端,逆转录酶又以第一条cDNA链为模板再合成第二第cDNA链,至此,完成逆转录全过程,合成双链cDNA。
逆转录现在已成为一项重要的分子生物学技术,广泛用于基因的克隆和表达。从逆转录病毒中提取的逆转录酶已商品化,最常用的有AMV逆转录酶。利用真核Mrna3’末端存在一段聚腺苷酸尾,可以合成一段寡聚胸苷酸(oligo(dT))用作引物,在逆转录酶催化下合成互补于mRNA的cRNA链,然后再用RNaseH将mRNA消化掉,再加入大肠杆菌的DNA聚合酶I催化合成另一条DNA链,即完成了从mRNA到双链DNA的逆转录过程。所得到的双链cDNA分子经S1核酸酶切平两端后接一个有限制酶切点的接头(linker),再经特定的限制酶消化产生粘性末端,即可与含互补末端的载体进行连接。常用的克隆载体是λ噬菌体DNA,如λgt,EMBL和Charon系列等。用这类载体可以得到包含104以上的转化子的文库,再经前面介绍的筛选方法筛选特定基因克隆。用这种技术获得的DNA探针不含有内含子序列。因此尤其适用于基因表达的检测。
(三)RNA探针
RNA探针是一类很有前途的核酸探针,由于RNA是单链分子,所以它与靶序列的杂交反应效率极高。早期采用的RNA探针是细胞mRNA探针和病毒RNA探针,这些RNA是在细胞基因转录或病毒复制过程中得到标记的,标记效率往往不高,且受到多种因素的制约。这类RNA探针主要用于研究目的,而不是用于检测。例如,在筛选逆转录病毒人类免疫缺陷病毒(HIV)的基因组DNA克隆时,因无DNA探针可利用,就利用HIV的全套标记mRNA作为探针,成功地筛选到多株HIV基因组DNA克隆。又如进行中的转录分析(nuclearrunontranscrip-tionassay)时,在体外将细胞核分离出来,然后在α-32P-ATP的存在下进行转录,所合成mR-NA均掺入同位素而得到标记,此混合mRNA与固定于硝酸纤维素滤膜上的某一特定的基因的DNA进行杂交,便可反映出该基因的转录状态,这是一种反向探针实验技术。
近几年体外转录技术不断完善,已相继建立了单向和双向体外转录系统。该系统主要基于一类新型载体pSP和pGEM,这类载体在多克隆位点两侧分别带有SP6启动子和T7启动子,在SP6RNA聚合酶或T7RNA聚合酶作用下可以进行RNA转录,如果在多克隆位点接头中插入了外源DNA片段,则可以此DNA两条链中的一条为模板转录生成RNA。这种体外转录反应效率很高,在1h内可合成近10μg的RNA产生,只要在底物中加入适量的放射性或生物素标记的NTP,则所合成的RNA可得到高效标记。该方法能有效地控制探针的长度并可提高标记物的利用率。
值得一提的是,通过改变外源基因的插入方向或选用不同的RNA聚合酶,可以控制RNA的转录方向,即以哪条DNA链以模板转录RNA。这种可以得到同义RNA探针(与mRNA同序列)和反义RNA探针(与mRNA互补),反义RNA又称cRNA,除可用于反义核酸研究外,还可用于检测mRNA的表达水平。在这种情况下,因为探针和靶序列均为单链,所以杂交的效率要比DNA-DNA杂交高几个数量级。RNA探针除可用于检测DNA和mRNA外,还有一个重要用途,在研究基因表达时,常常需要观察该基因的转录状况。在原核表达系统中外源基因不仅进行正向转录,有时还存在反向转录(即生成反义RNA),这种现象往往是外源基因表达不高的重要原因。另外,在真核系统,某些基因也存在反向转录,产生反义RNA,参与自身表达的调控。在这些情况下,要准确测定正向和反向转录水平就不能用双链DNA探针,而只能用RNA探针或单链DNA探针。
综上所述,RNA探针和cRNA探针具有DNA探针所不能比拟的高杂交效率,但RNA探针也存在易于降解和标记方法复杂等缺点。
(四)寡核酸探针
前述三种探针均是可克隆的,一般情况下,只要有克隆的探针,就不用寡核苷酸探针。在DNA序列未知而必须首先进行克隆以便绘制酶谱和测序时,也常应用克隆。克隆探针一般较寡核苷酸探针特异性强,复杂度也高,从统计学角度而言,较长的序列随机碰撞互补序列的机会较短序列少,克隆探针的另一优点是,可获得较强的杂交信号,因为克隆探针较寡核苷酸探针掺入的可检测标记基因更多。但是,较长的探针对于靶序列变异的识别能力又有所降低。对于仅是单个碱基或少数碱基不同的两序列,克隆探针不能区分,往往杂交信号相当。这既是其优点,又是其缺点。优点是当用于检测病原微生物时,不会因病毒或细菌DNA的少许变异而漏诊,缺点则是不能用于点突变的检测。这种情况下,通常要采用化学合成的寡核苷酸探针。
合成的寡核苷酸探针具有一些独特的优点:①由于链短,其序列复杂度低,分子量小,所以和等量靶位点完全杂交的时间比克隆探针短,如20nt的寡核苷酸探针在浓度为100ng/ml,靶序列为1~100pg、1kb片段或3×10-18~3×10-16mol/L时,达到最大程度的杂交只需10min,而用2kb的克隆探针在同样条件下达到完全杂交则需16h。②寡核苷酸探针可识别靶序列内1个碱基的变化,因为短探针中碱基的错配能大幅度地降低杂交体的Tm值。③一次可大量合成寡核苷酸探针(1~10mg),使得这种探针价格低廉,与克隆探针一样,寡核苷酸探针能够用酶学或化学方法修饰以进行非放射性标记物的标记。尽管克隆探针较特异,但通过细心筛选序列和/或选择相对长的序列(>30nt)亦可设计出非常特异的寡核苷酸探针。最常用的寡核苷酸探针有18~40个碱基,目前的合成仪可有效地合成至少50个碱基的探针。下面是筛选寡核苷酸针的一些原则。
①长18~50nt,较长探针杂交时间较长,合成量低;较短探针特异性会差些。②碱基成分:G+C含量为40%~60%,超出此范围则会增加非特异杂交。③探针分子内不应存在互补区,否则会出现抑制探针杂交的“发夹”状结构。④避免单一碱基的重复出现(不能多于4个),如-CCCCC-。⑤一旦选定某一序更符合上述标准,最好将序列与核酸库中核酸序列比较,探针序列应与含靶序列的核酸杂交,而与非靶区域的同源性不能超过70%或有连续8个或更多的碱基的同源,否则,该探针不能用。
按上述原则选出的探针会增加成功的机会,选定后进行合成与标记,并摸索合适的杂交条件。方法是制备几张点有特异靶DNA和不相关DNA的膜,各膜分别在不同温度下与探针杂交,特异靶DNA杂交信号强而非特异DNA不产生任何杂交反应的就是最适杂交温度。在进行点突变检测杂交的反应时,洗膜条件和温度物选择往往更为重要。所选漂洗条件必需使野生型靶DNA与探针产生强的杂交信号而突变型靶DNA则不产生杂交信号,这可以通过逐渐提高洗膜温度来完成。
寡核苷酸探针还有一个重要用途。在用于检测单个碱基差异时尚可采用一种称为寡核苷酸限制(oligonucleotiderestriction)的技术。该技术只有在突变点位于某一限制性内切酶识别位点时才有效。例如,镰刀状红细胞贫血是因β珠蛋白基因的第6个寡码子由GAG变成GTG,从而导致所编码氨基酸由酪氨酸变成缬氨酸。突变的β-珠蛋白功能异常,称作S珠蛋白,而野生型称为A珠蛋白,其基因型分别为βS和βA。恰好突变点A→T位于DelI的识别序列CT-NAG之内,这就为设计寡核苷酸限制实验创造了条件。方法是合成一个长40个碱基的寡核苷酸探针,其5’末端距突变碱基有11个碱基,该探针与βA基因的非编码链互补。将此探针的5’末端标记上32P。杂交方法采用液相杂交法,即在液相中将靶DNA变性解链,然后与探针退火,产生杂交体。如靶DNA为βA型,则两条链完全互补,并产生DdeI的酶切位点;如待检DNA为βS型,则所形成的杂交体中两条链在突变碱基处不配对,从而不能被DelI所识别。用DelI消化杂交DNA,显然βA会被切开而βS不被切开。
βADNA杂交体被切开后,5’端探针序列因只有8个碱基,与杂交链结合不紧而解离,从而产生游离的5’端标记8核苷酸单链。不被切开的βS杂交体尚可被另一个限制酶HinfI消化,该酶的识别位点紧靠DelI识别位点上游。βS杂交DNA经HinfI消化后,将释出探针DNA的5’末端3核苷酸小片段。βADNA杂交体因已无HinfI识别序列,故而不能被HinfI消化。这样βA和βSDNA经此寡核苷酸探针杂交和DelI及HinfI消化后,分别产生游离的8核苷酸(8nt)和3核苷酸(3nt)片段,它们可以经电泳分离后进行放射自显影而获证实。藉此策略,可轻易将各种β珠蛋白突变型鉴别开,如纯合野生型AA结果为仅有8nt片段,纯合突变型SS则仅可检出3nt片段,而杂合子AS型则两种片段均存在。
| 关于“分子杂交”的留言: | |
|
目前暂无留言 | |
| 添加留言 | |